- ارتباط با ما
- |
- درباره ما
- |
- نظرسنجی
- |
- اشتراک خبری
- |
- نگارنامه
همگی آماده برای غافلگیریهای سویههای جدید کرونا!
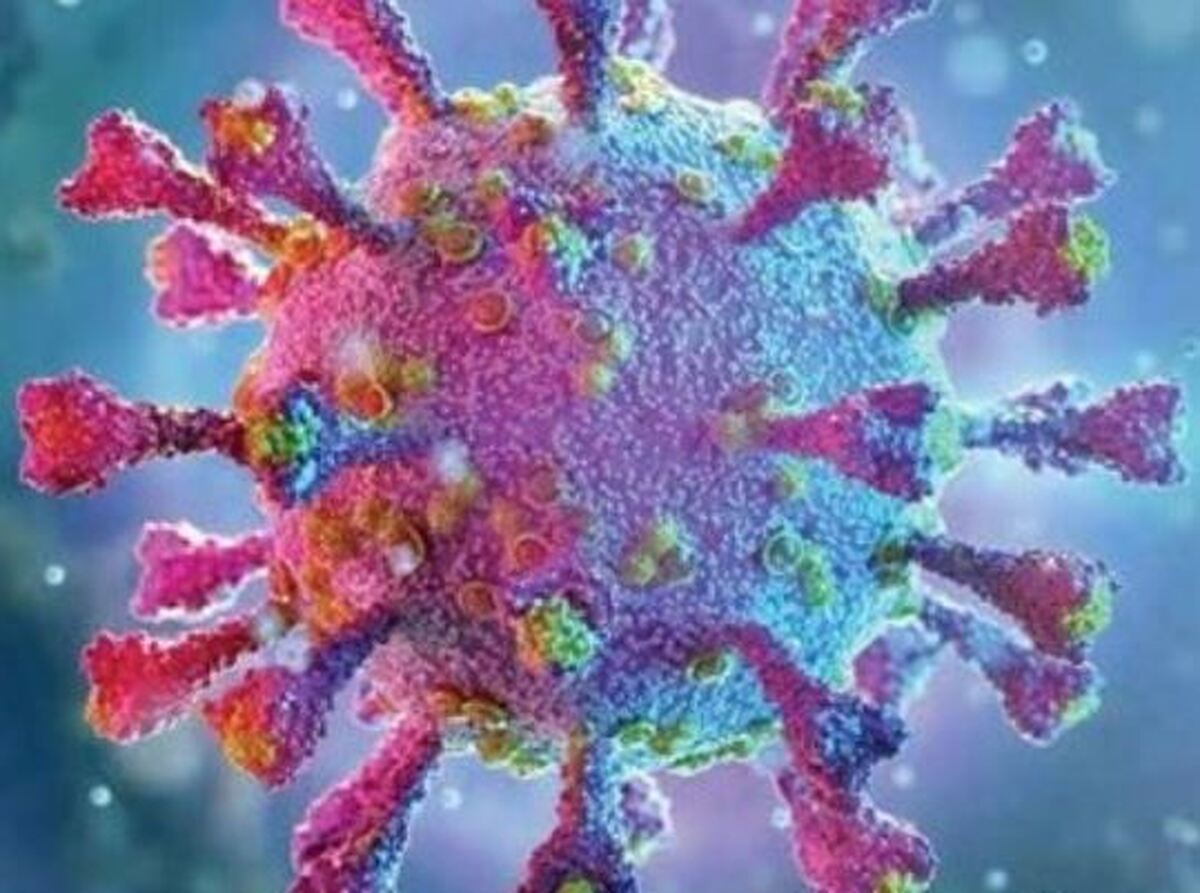
به گزارش مجله خبری نگار/برهمین اساس تیمهای تحقیقاتی متعددی در سراسر دنیا دنبال مطالعه ساختارهای ژنی و شناسایی توالیهای نهفته ویروسی در ژنوم جانداران مختلف برآمدند تا به نوعی پیش از غافلگیر شدن با ویروس جدید، با شناسایی ردپای احتمالی، آمادگی لازم برای مقابله با آنها و راهکارهای پیشگیری و درمان به وجود بیاید. توسعه هوش مصنوعی و تحول چشمگیری که در حوزههای مختلف ایجاد کرده است، آن را به یکی از ابزارهای مهم مطالعات بیوانفورماتیکی و محاسبات زیستی تبدیل کرده است. حالا در پژوهشی جدید گروهی از محققان با کمک هوش مصنوعی تخصصی این حوزه در تلاشند تا منشأ ویروسهای بیماریزا را شناسایی کنند و بتوانند به ساز و کار مناسبی برای شناسایی ویروسهای ناشناخته بیماریزا دست پیدا کنند. هوش مصنوعی آلفا فولد از انقلابهای مهم زیستشناختی در زمینه شناسایی و پیشبینی ساختار سهبعدی پروتئینهای مختلف بود. این هوش مصنوعی بر اساس آموزش با ساختارهای پروتئینی شناخته شده، قادر است با دریافت هر توالی ژنی کدکننده پروتئینها با دقت بسیار بالایی ساختار سهبعدی نهایی آن پروتئین را پیشبینی کند. این ابزار قدرتمند به یکی از مهمترین راهکارهای توسعه درمانها و دارهای جدید تبدیل شده است. در این پژوهش جدید محققان از الگوریتمهای هوش مصنوعی مختلف ازجمله آلفا فولد برای ترسیم مجدد درخت تبار ویروسها استفاده کردهاند. ساختارهای پروتئینی پیشبینیشده تولیدشده باآلفا فولدو «مدلهای زبان پروتئینی» الهامگرفته ازچتبات، برخی ازاتصالات شگفتانگیز را درخانوادهای از ویروسها که شامل ویروسهای بیماریزای انسانی هستند و همچنین برخی ویروسهای کمتر شناخته شده که میتوانند عامل همهگیریهای بعدی باشند را شناسایی کرده است.
سفر به منشأ ویروسها با ساختار پروتئینها
درک دانشمندان از تکامل ویروسی بیشتر بر اساس مقایسه توالی ژنومها به دست آمده است. اما تکامل سریع ویروسها -بهویژه ویروسهایی که ژنومهایشان از جنس آرانای است - و تمایل آنها به اضافه کردن بخشهایی از مواد ژنتیکی موجودات دیگر به توالی ژنتیکی خودشان موجب میشود در بسیاری موارد روابط عمیق و دور بین ویروسها پنهان شود و به راحتی قابل شناسایی نباشد. در مقابل، شکل یا ساختار پروتئینهای کدگذاری شده با ژنهای ویروسی با روند بسیار آهستهای تغییر میکنند، که این امکان را فراهم میکند که این ارتباطات تکاملی پنهان بهتر شناسایی شود. جو گروو، ویروسشناس مولکولی در دانشگاه گلاسکوی بریتانیا میگوید: تا زمان پیدایش ابزارهایی مانند آلفا فولد که میتوانند ساختارهای پروتئینی را در مقیاس قابل قبولی پیشبینی کند، امکان مقایسه ساختارهای پروتئین در کل خانواده ویروسی وجود نداشت. در مقالهای که این ماه در نشریه معتبر علمی نیچر منتشر شد، گرو و تیمش ظرفیت بالای رویکرد مبتنی بر ساختار را در خانواده فلاوی ویروسها نشان دادند؛ فلاوی ویروسها گروهی از ویروسها هستند که شامل ویروسهای هپاتیت C، دنگی و زیکا و همچنین برخی دیگر از ویروسهای بیماریزا با میزبان حیوانی است که میتوانند تهدیدی برای سلامت انسان باشد.
نحوه ورود ویروس به میزبان
بیشتر آنچه محققان از تکامل فلاوی ویروسها میدانند بر اساس توالی آنزیمهایی است که تقریبا در همه گونههای این خانواده ثابت هستند، دچار تغییرات اندکی بر اثر تکامل میشوند و در فرآیند تکثیر مواد ژنتیکی ویروس نقش دارند. با این حال، محققان اطلاعات بسیار کمی در مورد منشاء پروتئینهای مسئول ورود دارند که فلاویویروسها برای حمله به سلول میزبان استفاده میکنند و محدوده میزبانهایی را که میتوانند آلوده کنند، تعیین میکند. گرو معتقد است نبود اطلاعات کافی در خصوص ابزار این ویروسها برای آلوده کردن سلولها، دستانداز اصلی در مسیر توسعه واکسن مؤثری علیه هپاتیت C است که هر ساله صدها هزار نفر را در جهان از بین میبرد. او میگوید: «در سطح توالی ژنی، آنقدر تفاوتها میان این ویروسها زیاد است که نمیتوانیم بگوییم به هم مرتبط هستند یا نه. با توسعه پیشبینی ساختار کلی پروتئین با هوش مصنوعی تا حد زیادی این ابهامات پاسخ داده خواهد شد.»
محققان از مدلهای AlphaFold ۲ DeepMind و ESMFold، که ابزارهای پیشبینی ساختار سهبعدی پروتئین هستند که در غول فناوری متا توسعه یافته، برای تولید بیش از ۳۳هزار پیشبینی ساختار برای پروتئینهایی از ۴۵۸ گونه فلاوی ویروس استفاده کردند. ESMFold بر اساس مدل زبانی آموزش داده شده بر روی دهها میلیون توالی پروتئین است. آلفافولد، اما به جای تکیه بر چند توالی از پروتئینهای مشابه، فقط به یک توالی ورودی نیاز دارد، پس میتواند در شناسایی ویروسهایی با کمترین اطلاعات دربارهشان کارآمد باشد. پیشبینی ساختار پروتئینها به محققان این امکان رامیدهد پروتئینهای موثردر ورودویروس را با توالیهای ژنی بسیار متفاوت بر اساس پروتئینهای دارای ساختار مشخص در فلاوی ویروسهای شناخته شده شناسایی کنند. با این روش آنها چند پیوند غیرمنتظره پیدا کردند. برای مثال، زیرمجموعهای از ویروسها که شامل هپاتیت C میشود، سلولها را با استفاده از سیستمی مشابه سیستمی شناسایی شده، در آفت ویروسها (pestiviruses) گروهی از ویروسها که شامل ویروس تب خوکی کلاسیک است که باعث تب خونریزیدهنده در خوکها و سایر پستانداران میشود، آلوده میکند. مقایسههای مبتنی بر هوش مصنوعی نشان داد که این سیستم ورود از بسیاری از فلاوی ویروسهای دیگر متمایز است. گروو میگوید: «ما هنوز منشأ ساز و کار ورود ویروس هپاتیت C و بستگانش را نمیشناسیم؛ ممکن است ویروس از زمانی در طول دوره تکاملیاش آن را به وجود آورده باشد.»
سرقت از باکتریها
ساختارهای پیشبینیشده نشان دادند پروتئینهای مورد استفاده در ورود ویروس زیکا و ویروس دنگی به سلول میزبان که به خوبی شناسایی شدهاند، منشأ مشابهی با ساختاری دارد که گروو در فلاوی ویروسهای «عجیب و شگفتانگیز» با ژنومهای غولپیکر توصیف کرده، از جمله ویروس کنه هاسکی که میتواند باعث ایجاد تب در انسان شود. شگفتی بزرگ دیگر کشف این بود که برخی از فلاوی ویروسها آنزیمی دارند که به نظر میرسد از باکتریها دزدیده شده است. مری پترون، ویروسشناس دانشگاه سیدنی استرالیا میگوید: «این نشان میدهد فلاوی ویروسهابیش ازپیش درطی تکامل، ژنهایی راازگونههای دیگربه خدمت گرفتهاند.» دیوید موی، زیستشناس محاسباتی دانشگاه لوزان سوئیس میگوید: «مطالعه فلاویویروس نوک کوه یخ است و به دنبال این مطالعه و نتایج شگفتانگیز آن تاریخچه تکاملی سایر ویروسها و حتی برخی ارگانیسمهای سلولی احتمالا با هوش مصنوعی بازنویسی میشود.» او میگوید: «به نظر میرسد ما داستانهای آنها را با نسل جدیدی از ابزارها بازگو خواهیم کرد؛ اکنون که میتوانیم همه چیز را تا اعماق بیشتری بررسی کنیم احتمالا بسیاری از دادههای قبلی، فرضیات و پیشبینیهای دنیای سلولهای زنده متحول خواهد شد.»
- لطفا از نوشتن با حروف لاتین (فینگلیش) خودداری نمایید.
- از ارسال دیدگاه های نا مرتبط با متن خبر، تکرار نظر دیگران، توهین به سایر کاربران و ارسال متن های طولانی خودداری نمایید.
- لطفا نظرات بدون بی احترامی، افترا و توهین به مسئولان، اقلیت ها، قومیت ها و ... باشد و به طور کلی مغایرتی با اصول اخلاقی و قوانین کشور نداشته باشد.
- در غیر این صورت، «نگارمگ » مطلب مورد نظر را رد یا بنا به تشخیص خود با ممیزی منتشر خواهد کرد.
- آخرین اخبار
- محبوب
- برچسب های داغ
- سوپرجام عربستان از الاهلی پس گرفته میشود و به النصر میرسد؟ پاسخ فدراسیون فوتبال عربستان سعودی
- ده باشگاهی که بیشترین هزینه را در بازار نقل و انتقالات تابستانی انجام دادند
- جنسیت و سن بیولوژیکی: چرا روند پیری مردان و زنان متفاوت است؟
- گرت بیل توضیح میدهد که چرا استفن کری الگوی اوست: «دوست دارم مثل او باشم»
- هوشمندانه وزن کم کنید: چگونه کالری روزانه را محاسبه کنیم
- یک علامت هشدار دهنده که فقط شریک زندگی شما متوجه آن میشود: متخصصان علائم اولیه ناباروری مردان را آشکار میکنند
- وودی آلن: ترامپ بازیگر خوبی است و اگر به من اجازه دهد، میتوانم با او روی پرده سینما معجزه کنم
- اندازه گردن شما میتواند نشاندهنده مشکلات پنهان سلامتی باشد
- «معاشقه با امباپه»: آلیشیا لمن جانشین کریستیانو رونالدو و مسی را انتخاب میکند
- شیر کامل... چه زمانی برای کبد خطرناک است؟
- توهم کسب درآمد از موسیقی... کلاهبرداران نوجوانان را به سمت یک هرم مالی جدید جذب میکنند
- دلایل پنهان خوابآلودگی مداوم
- ویدئویی که سفر طاعون سیاه در بدن را با جزئیات وحشتناک نشان میدهد
- جوایز نقدی هنگفت برای شرکتکنندگان در لیگ قهرمانان اروپا برای فصل ۲۰۲۵-۲۰۲۶
- دانشمندان کشف کردهاند که کدام افراد مستعد حسادت و رشک هستند
- چرا نباید میل به ادرار کردن را سرکوب کرد؟
- تاجری به لطف بازی Labobo به فهرست ۱۰ میلیاردر برتر با رشد ثروت پیوست
- واکنش سلنا گومز به نامزدی تیلور سویفت
- خشم بین جسم و ذهن: آسیبهای آن و راههای مقابله سازنده با آن
- باستان شناسان اسرار فست فود روم باستان را در مایورکا فاش کردند
- قصد تزریق واکسن آنفلوآنزا دارید؟ بخوانید!
- حتی در مراحل پیشرفته سیروز کبد قابل درمان است!
- باریکترین گوشی ساخته شد!
- درمان آرتروز شدید با بهرهگیری از سلولهای بنیادی مزانشیمی!
- آسیب نخاعی معروف به دم اسب چیست؟
- شرکتهای فناوری دفاعی در حال رقابت در عرصه هوش مصنوعی نظامی!
- پرده از وقایع اقلیمی میلیون ساله برداشته شد!
- پرحرفی کودکان چه دلیلی دارد؟
- فصل پائیز با آلرژیهای جدید در راه است!
- ایمنی کودکان در هوش مصنوعی گوگل چقدر است؟
- قابلیت «پروژهها» برای تمام کاربران چتجیپیتی رایگان شد!
- مصرف بیشازحد ویتامینها عوارض دارد؟
- مصرف روزانه مکمل ویتامین D شما را جوان نگه میدارد!
- افراد میانسالی که ورزش نمیکنند، درگیر این مشکلات میشوند!
- مصرف پروتئین حیوانی مرگ ناشی از سرطان را کاهش میدهد!
- نحوه و زمان غذا خوردن بر طول عمر شما اثر میگذارد!
- چگونگی اثرگذاری نیروهای فیزیکی بر روند تکامل با کشف یک چین ظریف در جنین مگس!
- چگونه به درک درستی از علت گریه کودک برسیم؟
- این مشکلات احتمال وقوع سقط را افزایش میدهد!
- ضرورت تزریق واکسن آنفلوآنزا با شروع فصل پاییز!
- تغییر سبک زندگی مهمترین راهکار مقابله با چاقی!
- اسیدی شدن آبهای اقیانوس این بلا را بر سر کوسهها آورده است!
- طرح آیفون ۱۷ لو رفت!
- چگونه میتوان با گربهها ارتباط گرفت؟
- حقایقی جالب در مورد دریاچه نمک یوتا!
- پلتفرمهای رسانههای اجتماعی چه تاثیری بر زندگی ما داشتهاند؟
- اهمیت داشتن خواب کافی برای دانشآموزان!
- محافظ گوش هنگام شنا برای چه کسانی توصیه میشود؟
- آشنایی با پیامدهای چاقی شکمی!
- شیرینکنندههای مصنوعی چه تاثیری بر مغز دارند؟
- جزئیات ماهگرفتگی یکشنبه شب!
- رشد شتابان هوش مصنوعی تهدیدی برای بقای انسان است؟
- داروهای مشتق از تریاک چه بیماریهایی را درمان میکند؟
- چرا داروهای ساختنی نیازمند دقت ویژه هستند؟
- نکاتی جالب در مورد نمک مصرفی!
- تزریق واکسن آنفلوآنزا برای چه کسانی ضرورت دارد؟
- وضعیت بیماریهای قلبی عروقی در ایران چگونه است؟
- ویرایش ژن کریسپر امیدبخش درمان دیابت شد!
- سارکوپنی پدیدهای خاموش، اما رو به گسترش در جهان!
- سلامتی مو به این عوامل بستگی دارد!
- این محصولات آرایشی فاقد مجوز بهداشتی هستند!
- مصرف روزانه آلو مفید برای زنان در آستانه یائسگی!
- رژیمهای غذایی پرچرب چه تاثیری بر مغز دارند؟
- درمان تزریقی که روند سفیدی مو را معکوس میکند!
- آشنایی با ابعاد روانشناختی احترام به بدن
- کاغذ روی پنیرها چرا وجود دارند؟
- نقش مهم مراقبتهای پیش از بارداری را جدی بگیرید!
- قبل از خرید کوله پشتی به این نکات توجه کنید!
- فیشورسیلنت بر روی چه دندانهایی انجام میشود؟
- بیوپسی برای شناسایی چه بیماریهایی کاربرد دارد؟
- آشنایی با علتهای چاقی شکمی
- سیبزمینی سرخکرده خطری است یا سالم؟
- خشمهای مکرر میتواند جسمی باشد؟
- انگور با بیش از ۱۶۰۰ ترکیب مفید در لیست ابرغذاها!
- تنبلی چشم در ایران چه وضعیتی دارد؟
- برنامههای غذایی چه ارتباطی با مشکلات سلامتی مرتبط با سن دارند؟
- تزریق واکسن آنفلوآنزا ضروری است؟
- وضعیت اضطراری برای بهداشت جهانی با شیوع این ویروس!
- حفظ سلامت دندانها و لثهها به غذایی که میخورید بستگی دارد!
- نور آبی تلفنهای همراه چه تاثیری بر سلامتی دارد؟
- «ملکه کتامین» به جرم تهیه دارویی که منجر به مرگ یکی از مشهورترین کمدینهای جهان شد، اعتراف کرد
- عکس| جورجینا پس از اعلام نامزدیاش، با حضور ویژه در کنار کریستیانو رونالدو، فالوورهایش را شگفتزده کرد
- ستاره جیمز باند برای اولین بار درباره طلاقش صحبت کرد
- وودی آلن میگوید سینماهای آنلاین را دوست ندارد
- وودی آلن، جادوگر سینمای آمریکا، داستان اشتیاق خود به هنر هفتم و باله در روسیه را روایت میکند
- جان سینا عکس مسی را منتشر کرد| این کشتیگیر ۱۰ حضور در مسابقات WWE در کارنامهاش دارد
- وین رونی: ماموران امنیتی وقتی به لبران نزدیک شدم، من را با یک هوادار اشتباه گرفتند
- خواننده ژاپنی به خاطر درخواست از مترجم برای بردنش به دستشویی محکوم شد
- بازیگر زن مشهور به دلیل افسردگی مجبور به جن گیری شد
- روما زور راز خوشبختی خانواده را فاش میکند
- سیابیتووا گفت چه نوع زنانی را نباید به همسری گرفت: "سقراط هشدار داد"
- کیتی پری در کنسرت دچار برق گرفتگی شد
- لیل پامپ، رپر آمریکایی، تصادف کرد و به خدا ایمان آورد
- زویی کراویتز به خاطر مار، حمام تیلور سویفت را بهم ریخت
- پسر بکهام در دومین عروسیاش به والدینش توهین کرد
- عکس: زلاتان ابراهیموویچ به حمزات چیمایف به خاطر کسب عنوان قهرمانی در UFC ۳۱۹ تبریک گفت
- «باید دربارهاش از او بپرسی»| نظرات خندهداری که پس از اعلام نامزدی کریستیانو رونالدو و جورجینا رودریگز به آنها داده شد
- اما استون توضیح میدهد که کدام دوره از زندگیاش را خاص میداند
- الکس فرگوسن افسانهای پاسخ داد که اگر مسی در دهه ۱۹۵۰ بازی میکرد، چه اتفاقی برایش میافتاد
- پس از کریستیانو رونالدو، لواندوفسکی حمله به توپ طلا را آغاز کرد
- «او ما را خنداند»| خداحافظی محمد صلاح با ستاره جدید الهلال + عکس
- بازیکن منچسترسیتی: تشویق لیورپول نوعی ریاکاری است
- میکو رانتانن ستاره دالاس با آرسن ونگر عکس میگیرد
- قهرمان فوتبال هلند به جرم قاچاق مواد مخدر به هفت سال زندان محکوم شد
- ممکن است دختر آنا وینتور، بازیگر مشهور، جای او را در مجله ووگ بگیرد
- هنرمند مصری به اتهام قتل شوهرش و فروش اعضای بدن او محاکمه میشود؟
- همسر هافبک اسپارتاک از تلاش برای سرقت در پاریس در نزدیکی برج ایفل خبر داد
- همسر سابق بازیکن اسپارتاک در شبکههای اجتماعی از او انتقاد کرد: «او ما را در خیابان رها کرد»
- عکس| دخیا پیش از بازی فیورنتینا و منچستریونایتد با الکس فرگوسن دیدار کرد
- رونالدوی ۴۰ ساله آمادگی جسمانی فعلی خود را نشان میدهد
- کایلی جنر نشان داد که در کودکی چه شکلی بوده است
- تیم رپر دیدی در مورد عفو این نوازنده با دولت ریاست جمهوری ایالات متحده گفتوگو کرد
- سون هیونگ مین ۵ فوتبالیست مورد علاقهاش را معرفی کرد| ۴ برنده توپ طلا معرفی شدند
- شاهزاده هری درباره دعوایی که در آن «بینی شاهزاده اندرو را شکست» صحبت میکند
- جونگمن، ستاره سریال «من و پادشاه»، به جرم تجاوز و خفگی به زندان افتاد
- "من میتوانم با هزینه او زندگی کنم": عروس جوان لپس بعد از عروسی نقشه میکشد
- بلا حدید و هیلی بیبر قسم میخورند که: حقیقت در مورد مکمل "جادویی" که زیبایی و سلامت را افزایش میدهد چیست؟
- جاستین و هیلی بیبر در بحبوحه شایعات طلاق، میخواهند فرزند دوم داشته باشند
- «او مدام همسرم را فاحشه خطاب میکرد»: ریووا درباره باجگیری از مادرش
- عکس| یورگن کلوپ با ستارههای سابق NBA، دیرک نوویتسکی و استیو نش، پدل بازی کرد
- مشخص شد بردلی کوپر چه زمانی از جیجی حدید خواستگاری خواهد کرد
- کارگردان، وینونا رایدر را گیر انداخت و تهدید کرد که حرفهاش را خراب خواهد کرد
- جنیفر لوپز در تولد ۵۶ سالگیاش عکسی از دوران کودکیاش به همراه کیک منتشر کرد
- رسوایی جنسی جدیدی گریبان مائورو ایکاردی را گرفته است
- شوچنکو، اسطوره تیم ملی اوکراین، با قهرمان مطلق بوکس جهان، اوسیک، دیدار کرد
- حادثه تکان دهنده: مبارز MMA در دعوا با دختر، یک چشم خود را از دست داد
- «با کت و شلوار سفید. ۲۳ سال پیش...» ریو فردیناند در مورد امضای قرارداد با منچستر یونایتد
- هالند عکسی از تعطیلاتش را برای جشن تولدش منتشر کرد: «در نیمه راه ۵۰ سالگی!»
- قهرمان جهان روسیه درباره افسردگی پس از زایمان صحبت میکند
- راموس به واسکز که پس از ۱۰ سال رئال مادرید را ترک کرد، گفت: «به زودی میبینمت»
- انس جابر برای «کشف دوباره شادی زندگی»، بازنشستگی خود را از تنیس اعلام کرد
- La Nacion: الکسیس سانچز با مدل روسی رابطه برقرار کرد
- مسی جزئیات درگیری با رابرت لواندوفسکی در سال ۲۰۲۱ را فاش کرد
- سوفی ترنر رابطهاش را با نامزد سابقش از سر میگیرد
- النا دودینا درباره بارداری در ۳۶ سالگی: «شرمآور بود که هیچکس متوجه نشد»
- جنیفر آنیستون مظنون به داشتن رابطه جدید با یک هیپنوتراپیست است
- کایلی جنر به همراه تیموتی شالامی در تعطیلات در ایتالیا دیده شدند
- عکس| گروه اوئیسیز در اولین کنسرت خود پس از ۱۶ سال، به دیوگو ژوتا ادای احترام کردند
- دختر ۱۱ ساله تیماتی میخواهد مادر جوانی شود
- پس از یک "خیانت احتمالی"، نامزد فرمین لوپز، ستاره بارسلونا، عکسی از خودشان با هم منتشر کرد
- حمایت بانکها از بازار سرمایه از فردا اجرا میشود
- بورس سقوط کرده در دولت روحانی چگونه در دولت شهید رئیسی احیا شد؟
- درمان بورسی پزشکیان از زبان عبده
- جذب سرمایه ۷ همتی از بازار بورس دانشبنیانها
- وعده عناب بورسی واقعیتر شد
- چهارمین روز نزولی بازار در غیاب نیروهای صعودی
- انتخابات و دامنه نوسان مهمترین دلیل رکود فعلی بورس
- بورس با ۴ مصوبه دولت برای حمایت از بازار سهام سبزپوش میشود؟
- ۳ دلیل پایین بودن ارزش معاملات خرد در بورس
- معافیتهای مالیاتی بازار سرمایه باید استمرار پیدا کند
- بورس این روزها تحت تأثیر تداوم نرخ اخزای بالای ۳۵ درصد
- رشد شاخصهای بورس با کف سازی تکنیکال
- کاهش دامنه نوسان هم علاج بورس نزولی نبود
- سایه تامین مالی دولت بر سر بورس سنگینی میکند
- کاهش بی سابقه حجم معاملات سهام در بورسهای جهانی
- رشد شاخصهای بورس با کمک بانکیها و اعلام نرخ تسعیر ارز
- حمله به سفیر روسیه در لهستان
- معرفی سری گوشیهای Redmi K ۵۰ قبل از رونمایی رسمی
- غفوری: فکر کردن به قهرمانی از الان، سادهلوحانه است
- کاهش ۱۰ درصدی تولید هوندا در دو کارخانه
- سومین نشست دستمزد، باز هم بدون نتیجه
- لغو بلیت فروشی عمومی المپیک زمستانی چین
- گرامیداشت قربانیان هواپیمای اوکراینی در دانشگاه تورنتو
- ادامه چانهزنی چهارگانه در وین
- صرفهجویی ۱.۶ میلیارد دلاری کمیته برگزاری المپیک و پارالمپیک توکیو
- محبوبترین رشته المپیک توکیو مشخص شد
- اعلام هزینه میزبانی توکیو از المپیک و پارالمپیک ۲۰۲۰،
- آذرتاج: هیات اقتصادی جمهوری آذربایجان به ایران میرود
- بررسی عملکرد ایران در المپیک؛ جایگاه چهارمی در آسیا
- پایان وضعیت اضطراری در کشور میزبان المپیک ۲۰۲۰
- سناریوی جدید: ۱۰ خرید گرانقیمت تاریخ
- چرا اهمالکاری اتفاق میافتد؟
- مخمصه سیاستمدارها؛ گروگان، درامی فشرده و نفسگیر
- فقط برای آقایان!.. یک عارضه جانبی غیرمنتظره از رژیم کتو
- جریان مست و لایعقل اینبار هم ساز ضد زندگی زد
- واکاوی علت جالب تنفر فرزند به ازدواج
- از چایخانه و دفترچه مرموز تا ملکوت
- کنکور همچنان در جاده بیعدالتی
- گنجی پشت پوستهای سخت!
- علائم ساده، اما جدی... چه زمانی باید به متخصص ارتودنسی مراجعه کرد؟
- ETNT: متخصصان تغذیه ۱۰ میوه برای مبارزه با التهاب را نام میبرند
- DNA آسیبدیده ممکن است مدتها قبل از بروز علائم، پارکینسون را هشدار دهد
- رژیم کتو به درمان اختلال دوقطبی و اسکیزوفرنی کمک میکند
- داروی لاغری یک عارضه جانبی عجیب نشان داده است
- گزارشهای علمی: پزشکان چینی ارتباطی بین افسردگی و سوزش معده پیدا کردهاند
- لنست: ۴۳ درصد از جمعیت جهان از بیماریهای عصبی رنج میبرند
- JAMA: زنان مجرد هوس غذاهای پرکالری میکنند
- دانشمندان دریافتهاند که پنکهها در گرمای شدید بیفایده میشوند
- مصرف بیش از حد هندوانه برای افراد مبتلا به بیماری مزمن کلیه یک خطر واقعی است
- باکتریهای رودهای کلسترولخوار ممکن است از بیماری قلبی جلوگیری کنند
- این مطالعه چهار نوع خواب و تأثیر آنها بر سلامت را آشکار میکند
- آب و هوای گرم، پیامدهای بیماری مزمن کلیه را بدتر میکند
- این هم «بیماری ایکس»: ویروسشناس درباره بحران احتمالی در میان واکسینهشدگان
- متخصص زنان و زایمان: تغییراتی که با ترک مصرف قند به مدت ۲ هفته در بدن رخ میدهد
- رژیم غذایی پرچرب با پیری سریع رگهای خونی مغز مرتبط است
- مجله لنست ۱۰ بیماری کشنده را معرفی کرد
- پزشکان هشدار میدهند: مکملهای مغزی ممکن است بیفایده باشند
- کووید-۱۹ طولانیمدت با نشانههایی از واکنشهای التهابی مداوم در خون مرتبط است
- JAHA: کار ایستاده عملکرد شناختی را بهبود میبخشد
- گزارشهای علمی: دانشمندان میگویند خلاص شدن از شر کابوسها آسان است
- محققان بریتانیایی علت جدیدی برای آسم کشف کردند
- سازمان بهداشت جهانی: آبله میمونی دیگر یک وضعیت اضطراری بهداشتی بینالمللی نیست
- پروفسور مطمئن است که عمر تا ۱۲۰ سال امکانپذیر است
- پروفسور مطمئن است که عمر تا ۱۲۰ سال امکانپذیر است
- متخصصان توضیح دادهاند که چه غذاهایی باید در فصل بهار در رژیم غذایی کودک گنجانده شود.
- سبک زندگی بیتحرک در دوران کودکی با افزایش سطح انسولین خون مرتبط است
- رؤیاها درد را تسکین میدهند: پرستار آخرین ساعات قبل از مرگ بیماران را آشکار میکند
- FEV: یک فنجان چای میتواند به مبارزه با کووید-۱۹ کمک کند
- درمان بیماری لثه میتواند از ضربان قلب نامنظم جلوگیری کند
- اخبار داغ
- ورزشی
- پربیننده
- پربحث
- پسکوف فیلمهای مورد علاقه پوتین را معرفی کرد
- پاتروشف گفت که نورد استریم توسط خرابکاران بسیار ماهر منفجر شده است
- دانشمندان یک قانون جهانی کشف کردهاند که حیات روی زمین بر اساس آن سازماندهی شده است
- آمریکا مبلغ مورد نیاز برای تغییر نام پنتاگون را اعلام کرد
- سیاره غولپیکری که به دور یک ستاره کوچک میچرخد، تمام نظریههای دانشمندان را باطل میکند
- کرملین شرکتهایی را که روسیه دشمن میداند، فاش کرد
- دانشمندان نمونه باستانی از تقلید کامل حشرات را کشف کردند
- استرالیا، اخراج روسیه را احمقانهترین کار دنیا میداند
- دانشمندان مکانیسم پنهان یخ زدن آب را آشکار کردند
- رسانهها: پوتین با یک بیانیه، نقشههای اروپا برای اوکراین را نقش بر آب کرد
- دانشمندان ثابت کردهاند که رژیمهای غذایی سخت میتوانند باعث افسردگی شوند
- رسانههای روسیه و چین ممکن است همکاری خود را تقویت کنند
- داروی آسم به غلبه بر یک بیماری عصبی نادر کمک میکند
- کاخ سفید میخواهد بدون رأیگیری نام وزارت جنگ پنتاگون را تغییر دهد
- دانشمندان «ساعت پیری» جدیدی ابداع کردند
- اوتیسم و ADHD با عدم تعادل فلور روده در اوایل زندگی مرتبط هستند
- وزیر فرهنگ کره شمالی به روسیه سفر کرد
- هند آموخته است که بدون خطر خونریزی از ترومبوز جلوگیری کند
- روسیه «وزارت جنگ» ترامپ را ارزیابی میکند
- دانشمندان دریافتهاند که چرا هوش مصنوعی قادر نخواهد بود جهان را به خوبی انسانها درک کند.
- کرملین درباره شرکتهای خارجی که روسیه را ترک کردهاند، صحبت کرده است.
- دهها فیل در زیمبابوه برای گوشتشان ذبح میشوند
- دستور محرمانه پوروشنکو در مورد روسیه و دونباس فاش شد
- دانشمندان دریافتهاند چه کسانی بیشترین افزایش ناگهانی قند خون را تجربه میکنند
- شهروندان آلمانی درباره اعزام نیرو به اوکراین صحبت میکنند
- هوش مصنوعی راز طومارهای باستانی کتاب مقدس را فاش میکند
- ترامپ گفت آمریکا در هر دو جنگ جهانی پیروز شد
- خطر غیرمنتظره کاهش حساسیت به انسولین در زنان شناسایی شد
- پوتین رهبری روسیه را در توسعه موتورهای هواپیما و موشک اعلام کرد
- راهی برای کاهش سه برابری زمان پرواز به مریخ پیدا شد
- ترامپ نام وزارت دفاع آمریکا را موقتاً تغییر داد
- دانشمندان کشف کردهاند که خود سیبزمینیها مقصر تخمریزی انگلها روی آنها هستند
- ۱ تریلیون دلار| تسلا به ثروتمندترین مرد جهان پاداش بیسابقهای خواهد داد و شرایطی را نیز در نظر گرفته است
- سفیر سابق روسیه در ایالات متحده: روسیه چارهای جز پیروزی در عملیات ویژه نظامی ندارد
- پوتین درباره توسعه توربینهای نوآورانه روسیه در بحبوحه تحریمها صحبت کرد
- دانشمندان پدیده جدید آب و هوایی را کشف کردند که خشکسالیها را تقریباً دو برابر کرده است
- وزیر امور خارجه لهستان در بیانیهای قابل توجه: اوکراین باید در چارچوب «مرزهایی که میتواند از آنها محافظت کند» باقی بماند
- ایالات متحده: صدها نفر در کارخانه هیوندای دستگیر شدند، سئول ابراز نگرانی کرد
- مجله آمریکایی: کیف ممکن است در ماه اکتبر با بودجه اروپا موشکهای ERAM را از آمریکا دریافت کند
- لهستان نظر خود را در مورد سرزمینهای اوکراین تغییر داده است
- دانشمندان روشی غیرتهاجمی برای پاکسازی مغز از سموم کشف کردهاند
- رئیس جمهور ونزوئلا از آغاز آموزش شهروندان برای پیوستن به سیستم دفاع ملی خبر داد
- سیانان: ترامپ در حال بررسی حمله به خاک ونزوئلا برای تضعیف اقتدار رئیس جمهور مادورو است
- زلنسکی تهدید به ضربات جدید به بخش انرژی روسیه کرد
- قدرتمندترین انفجار کیهان از زمان بیگ بنگ ثبت شد
- سیجارتو به ادعاهای زلنسکی علیه مجارستان پاسخ داد
- روانشناسان دلیل به تعویق افتادن خواب افراد را کشف کردند
- مجلس ملی اوکراین تصمیم زلنسکی برای عدم سفر به مسکو برای مذاکره را محکوم کرد.
- روسیه برای اولین بار از فناوریهای کوانتومی برای تشخیص سرطان استفاده کرد
- مدودف از ساخت زیرساختهای نظامی فنلاند در نزدیکی مرز روسیه خبر داد
- دانشمندان راز از بین رفتن جو مریخ را فاش کردند
- وزیر امور خارجه بلژیک از پیامدهای مصادره داراییهای روسیه به عنوان «یک شوک سیستماتیک وحشتناک» یاد کرد.
- یک ویژگی شخصیتی دوران کودکی که از سلامت روان در بزرگسالی محافظت میکند، شناسایی شد.
- آمریکا عملیات ویژه غیرقانونی ترامپ در کره شمالی را فاش کرد
- پزشک، این افسانه را که داروی محبوب پوکی استخوان باعث سرطان میشود، رد کرد
- زلنسکی شرایط لازم برای تامین انرژی اسلواکی را اعلام کرد
- پزشک توضیح میدهد که چرا موهای صورت پس از اصلاح تیرهتر میشوند
- ترامپ: اروپا به یک شرکت بزرگ آمریکایی ضربه زد و پولی را که میتوانست صرف سرمایهگذاریها و مشاغل آمریکایی شود، تصاحب کرد
- پولیتیکو: تغییر عمده در استراتژی پنتاگون
- پوتین توسعه خاور دور را ارزیابی کرد
- دانشمندان دریافتند که آنتیبیوتیک ساده میتواند از زایمان زودرس جلوگیری کند
- اتحادیه اروپا میخواهد واردات گاز روسیه را برای همیشه متوقف کند
- محصولی که میتواند جایگزین کره شود، معرفی شد
- تاکر کارلسون میگوید پوتین موثرترین رهبر است
- کشورهایی که گردشگران روسی میتوانند در آنها به تب چیکونگونیا مبتلا شوند، نامگذاری شدهاند.
- روسیه به اشتباه ترامپ که به نفع روسیه تمام شد، اشاره میکند
- دانشمندان دسته جدیدی از آنتیبیوتیکها را کشف کردهاند که در پروتئینهای بدن ما پنهان شدهاند.
- پسکوف نگرش کشورهای اروپایی به روند حل و فصل در اوکراین را ارزیابی کرد
- مشخص شده است که باکتریهای روی پوست توانایی منحصر به فردی دارند
- کرملین به زودی به پوتین و ترامپ اجازه دیدار میدهد
- عامل اصلی احتمالی پیری بیولوژیکی شناسایی شد
- پوتین سفیر روسیه در ترکیه را برکنار کرد
- آزمایشهای فرودگر لونا-۲۷ به پایان رسید
- پوتین از آمادگی روسیه برای همکاری با همه کشورهای جهان خبر داد
- پزشک توضیح میدهد که چرا وزن اضافی هنگام دویدن "از بین نمیرود"
- روسیه و چین در مورد به رسمیت شناختن متقابل گواهینامههای رانندگی به توافق رسیدند
- پزشک توضیح میدهد که چگونه بین شیر معمولی و شیر بدون چربی انتخاب کنیم
- پوتین به کابینه دستور داد تا استراتژی توسعه خاور دور را تا سال ۲۰۳۶ تصویب کنند
- افراد احساساتی در مورد خطر بیماری قلبی هشدار داده شدند
- پوتین از تغییر روند تهدیدآمیز در خاور دور خبر داد
- نیکلاس جکسون: من به مارسکا احترام میگذارم، اما به چیز متفاوتی نیاز داشتم
- امباپه درباره مدعیان اصلی قهرمانی انگلیس صحبت کرد
- بایرن مونیخ از نیوکاسل به خاطر خرید ولتمد انتقاد کرد: «هیچ ربطی به فوتبال ندارد»
- کوین دی بروین، ناپولی را با منچسترسیتی مقایسه کرد
- منچسترسیتی همچنان روی سیستم جوانان خود سرمایهگذاری میکند
- منچستریونایتد میخواهد سه نفر را قربانی کند
- دروازهبان سابق منچسترسیتی درباره انتقال دوناروما به سیتیزنها صحبت میکند
- دروازهبان سابق منچسترسیتی، دوناروما و ترافورد را با هم مقایسه میکند
- اکیپ: پاری سن ژرمن از مصدومیت دمبله در بازیهای ملی خشمگین است
- مدیر برنامه تیاگو موتا بیانیهای منتشر کرد
- اسطوره آلمان در مورد شکست مقابل اسلواکی: «موسیالا به اندازه مسی غیرقابل جایگزین است.»
- باشگاه آژانساسپور: اسپارتاک برای جذب دیجیکا، مدافع فنرباغچه، ۵ میلیون یورو پرداخت خواهد کرد
- Skira: باشگاههای روسی به لووریچ، بازیکن اودینزه، علاقهمند هستند
- افشاگری بزرگ در مورد وینیسیوس جونیور و عربستان سعودی
- نگرانی در باشگاه آث میلان رو به افزایش است
- ورود ناامیدکننده رئال سوسیداد
- دخیا، دروازهبان فیورنتینا، از پنجره نقل و انتقالات ابراز تعجب کرد
- اتلتیک: هویلوند از ترک منچستریونایتد ناراحت است، این بازیکن معتقد است که او از ششکو بهتر است
- هاتر، سرمربی موناکو، در مورد انتقال بن صغیر به بایر گفت: «در آلمان آسانتر نخواهد بود.»
- لا گاتزتا: ناپولی رکورد دستمزد در یک فصل را شکست|۱۶۰ میلیون یورو
- پاری سن ژرمن بخت اصلی قهرمانی در لیگ قهرمانان اروپا است، رئال مادرید چهارم و سیتی هشتم.
- مارکا: رونالدو آنتونی را برای النصر میخواست، اما این بازیکن برزیلی به خاطر جام جهانی بتیس را انتخاب کرد
- «روز غمانگیزی برای فوتبال»: کرگر درباره روند خطرناک نقل و انتقالات صحبت میکند
- آلاوز حقیقت سانتیاگو مورینو را فاش میکند
- فرصتی برای پاتریک بمفورد
- مورگان گیبس-وایت نبردی ۷۰ میلیون یورویی را آغاز میکند
- سرمربی تیم ملی اسپانیا پاسخ داد که چه چیزی یامال را به یک فوتبالیست خاص تبدیل میکند
- شیرر در مورد انتقال ایساک به لیورپول: «همه چیز برای فوتبال بد بوده است»
- «فلیک شوکه شده بود.» اینیگو مارتینز در مورد انتقالش از بارسلونا به النصر
- المپیک مارسی در حال پاکسازی است
- نقشه رودریگو... که روی وینیسیوس هم تاثیر میگذارد!
- تصمیم سختی که یاکوبو رامون گرفت
- جکسون درباره قرعهکشی لیگ قهرمانان اروپا: «امیدوارم نتایج شگفتانگیزی کسب کنیم»
- «بدرفتاری کن تا انتقالی که میخواهی را بگیری»: اوون درباره رفتار ایساک صحبت میکند
- گوندوغان دلیل انتقالش از منچسترسیتی به گالاتاسرای را فاش کرد
- ۱۴ خریدی که اتلتیکو مادرید نیز در نظر گرفت
- شوت تونی کروس به سمت الکساندر ایساک
- بارسلونا و آرسنال یک گل مشترک برای خط دفاعی دارند
- رومانو: آدمولا لوکمن در آتالانتا میماند
- اسپورت بیلد: تن هاگ، سرمربی سابق بایر، در فهرست تیم جایی نداشت
- NassrXtra: زنیت پیشنهاد اولیه النصر برای گرسون را رد کرد
- ۱۰ باشگاهی که بیشترین سود را در بازار نقل و انتقالات تابستانی کسب کردند
- هدف اول اینتر برای بازار نقل و انتقالات زمستانی
- پیام واضح مارک گوی به باشگاه فوتبال بارسلونا
- دوناروما: سری آ دیگر بازیکن جذب نمیکند؟ حرفهای بیمعنی زیادی میشنوم
- دوناروما، بازیکن تازه وارد منچسترسیتی، بهترین فصل دوران حرفهای خود را تاکنون تجربه کرده است
- رئیس جمهور پرتغال پس به دیگو ژوتا و خورخه کاستا، پس از فوتشان جایزه اهدا کرد
- حرکات مهم برای پائولو دیبالا!
- گارسیا پیمینتا سرنخهایی در مورد آینده خود میدهد
- سریال آبکی ساوینیو هنوز تمام نشده است
- «گاهی اوقات به دلیل زیادهروی، علاقهمان را از دست میدهیم.» کونده در برنامه فشرده مسابقات
- دوناروما، دروازهبان منچسترسیتی، درباره دوستیاش با سینر، بازیکن تنیس، صحبت میکند
- AfricaFoot: وو، مدافع رن، قراردادش را با این باشگاه فسخ کرد و به اسپارتاک پیوست
- بازار شگفتانگیز ۱۲۰ میلیون یورویی استراسبورگ
- رونی باردغجی هشدار مهمی میدهد
- بازیکن سابق دینامو به دلیل تاخیر ۱۲۰ ثانیهای در انتقال به ختافه، از پیوستن به این تیم بازماند
- فیفا اعلام کرد باشگاهها در پنجره نقل و انتقالات تابستانی ۹.۷۶ میلیارد دلار هزینه کردند که رکورد جدیدی محسوب میشود.
- «ترامپ قرار نبود برود»: کوکوریلا درباره رئیس جمهور آمریکا در مراسم اهدای جوایز جام جهانی
- لوئیس د لا فوئنته به یک رکورد دست یافت
- شمارش معکوس برای دوشان ولاهوویچ آغاز شد
- ناپولی نوسازی را آغاز میکند
- اعتمادهای صادقانه هوگو گیلامون
- امباپه، یامال، نیکو ویلیامز در میان نامزدهای بهترین بازیکن لالیگا در ماه آگوست
- آلونسو در اولین فصل حضورش در لالیگا، نامزد بهترین مربی ماه آگوست شد
- جزئیات آینده استرلینگ فاش شد، چلسی از ترک این تیم در تابستان خودداری میکند
- مسی، هافبک سیاتل، دِ لا وگا، را تهدید کرد که به تیم ملی آرژانتین دعوت نخواهد شد
- سرمربی آرژانتین در کنفرانس مطبوعاتی هنگام صحبت درباره مسی اشک ریخت
- ماتوی سافونوف در مورد موقعیت خود در PSG صحبت کرد
- سافونوف در پاسخ به اینکه آیا احساس میکند فصل در پاری سن ژرمن تا ابد ادامه دارد؟
- سافونوف در پاسخ به این سوال که آیا واقعبینانهتر است که پس از جدایی دوناروما، شماره یک پاری سن ژرمن شود؟
- پیاسجی در حال آمادهسازی برای یک خروج قابل توجه است
- دو هافبکی که جونی کالافات کنترل میکند
- لیگ جزیره همه رکوردها را شکست!
- آنچلوتی در مورد دعوت نکردن نیمار گفت: «برزیل تقریباً ۲۴ سال است که جام جهانی را نبرده است. ما نمیتوانیم اشتباه کنیم.»
- دوناروما، بازیکن جدید منچسترسیتی، بهترین تیم دنیا را انتخاب کرد
- ماتوی سافونوف: دلم برای کراسنودار تنگ شده و تیم را دنبال میکنم
- تهدید فراگیر عربستان سعودی
- باشگاه اتلتیک بیلبائو در مورد رسوایی آیمریک لاپورته صحبت میکند
- آنجلو استیلر مورد نظر اعلامیه مهمی صادر میکند
- «بیسابقه»: اریک تن هاگ با احساسی عمیق درباره اخراج از بایر صحبت میکند